
R语言furrr包进行并行运算
背景
前一段时间用R进行样本量计算模拟时,发现了一个小问题,R默认只会调用CPU的单核心来进行统计计算,一般情况下倒是也不会花费太多时间,不过当计算量巨大时,个人电脑算的也太慢了,于是就有了这一篇文章,用来记录自己的学习过程。
操作
for循环
假设我们需要生成200个服从标准正态分布*N~(0,1)*的随机数,并重复生成2000次,共计生成,先写一个for循环如下:
normal_data <- function(i){
a <- rnorm(200) #随机生成200个服从N(0,1)的随机数。
b <- data.frame(x = a) #将随机数存储为数据库中
return(b)
}
data_1 <- data.frame()
random_seed <- 20230704 #设定随机种子
set.seed(random_seed)
for (i in 1:2000){
a <- normal_data(i)
data_1 <- rbind(data_1,a)
}将其保存到R的工作目录下,命名为orgin.r文件。
future并行运算
使用furrr包运行该代码:
library(furrr)
future::availableCores() # 查看你的主机有多少个核心
plan(multisession(workers = 4)) #选择4个核心作为并行运算
normal_data <- function(i){
a <- rnorm(200)
b <- data.frame(x = a)
return(b)
}
random_seed <- 20230704
set.seed(random_seed)
data1 <- furrr::future_map_dfr(1:2000,normal_data,.options = furrr_options(seed = TRUE)) #设置option开启随机种子。将该代码保存在R的工作目录下,命名为furrr.r文件。
比较运行时间
system.time(source("orgin.r"))
system.time(source("furrr.r"))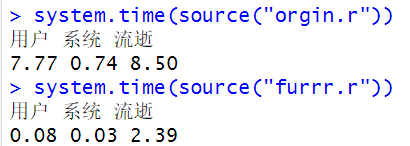
此时应该可以看出采用并行运算明显的缩短了程序运行的时间,完毕!

